上期小编给大家分享了eDNA在动物食性方面的研究应用。今天,小编给大家带来的是如何利用eDNA技术对水体进行研究,同时,希望这几篇文章能给有这方面研究的老师带来一些思路。
案例一:1.被动eDNA采样有利于生物多样性监测和稀有物种检测

文章题目:Passive eDNA sampling facilitates biodiversity monitoring and rare species detection
发表期刊:Environment International
图1(a)两种采样方法检出鱼类物种数量的累计曲线;
(b)两种方法在各采样点上检出物种数的成对比较
发表时间:2024.04
引物信息:
12S rRNA
Tele02_F:5′-AAACTCGTGCCAGCCACC-3′
Tele02_R:5′-GGGTATCTAATCCCAGTTTG-3′
摘要
环境DNA(eDNA)技术为生物监测带来了革命性的变化,但在水样处理方面仍存在挑战。被动eDNA采样器(PEDS)代表了一种可行的替代方法——基于水过滤的主动eDNA富集方法,但PEDS在调查生物多样性和复杂的自然水体中的有效性尚不清楚。本研究以长江终段和黄海沿岸的27个采样点为研究对象,采用过滤法和玻璃纤维过滤法采集了水中浸泡1d的PEDS样本,并进行了鱼类生物多样性的eDNA宏条形码分析和濒危水生哺乳动物长江江豚的定量PCR(qPCR)。最终通过eDNA宏条形码检测到98种鱼类。两种eDNA取样方法均捕获了相当的本地物种丰富度,并揭示了河流和海洋地点之间鱼类群落的空间变异和群落分区的基本相似。值得注意的是,只有在PEDS收集的5个位点的eDNA的宏条码中检测到长江江豚。此外,物种特异性qPCR显示,与过滤相比,PEDS捕获了更多的位点、更多的数量和更高的检测概率。本研究结果证明了PEDS在调查鱼类生物多样性方面的能力,并支持通过PEDS连续采集eDNA在捕获自然水域的低丰度和“短暂”停留的物种方面比瞬时采样更有效。因此,PEDS方法可以更高效和方便地对生物多样性监测和稀有物种进行检测。
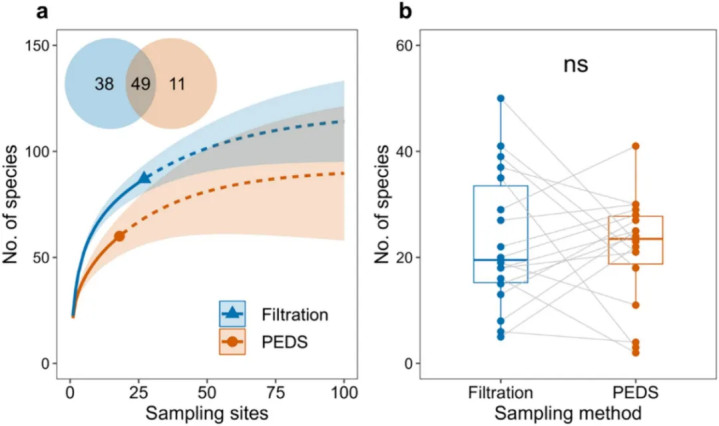
图1(a)两种采样方法检出鱼类物种数量的累计曲线;
(b)两种方法在各采样点上检出物种数的成对比较
案例二:2.eDNA宏条形码显示了在一个海洋生物多样性热点区域内,不同深度的真核生物群的高度多样化

文章题目:eDNA metabarcoding shows highly diverse but distinct shallow, mid-water, and deep-water eukaryotic communities within a marine biodiversity hotspot
发表时间:2024.07
引物信息:COI
mICOIintF-XT:5′-GGWACWRGWTGRACWITITAYCCYCC-3′
dgHCO2198:5′-TAIACYTCIGGRTGICCRAARAAYCA-3′
摘要
随着人类活动的影响继续从浅海向更深的海洋层移动,描述来自深层的真核生物组合如何与浅海生态系统相比较是至关重要的。环境DNA(eDNA)宏条形码作为一种工具,在探索遥远的深海区域时可以克服许多障碍。本研究分析了32对来自公认的海洋生物多样性热点地区的浅层(

图2 不同深度OTU丰度及分类的Venn图
案例三:3.eDNA宏条形码揭示了西部珊瑚海上浮游带中甲藻群落的生物多样性和深度分层模式

文章题目:eDNA metabarcoding reveals biodiversity and depth stratification patterns of dinoflagellate assemblages within the epipelagic zone of the western Coral Sea
发表期刊:BMC Ecology and Evolution
发表时间:2024.07
引物信息:18S rRNA
V418SNextFor:5’-TCGTCGGCAGCGTCAGATGTGTATAAGAGACAG
V418SNextRev:5’-GTCTCGTGGGCTCGGAGATGTGTATAAGAGACAG
摘要
鞭毛藻在海洋生态系统中发挥重要作用,但也可能通过引起有害的藻华(HABs)对人类和生态系统健康造成危害。本研究使用18S rRNA V4扩增子的宏条形码来评估在西珊瑚海的三个站点,在150米深度的水柱中甲藻的多样性和结构。此外,该研究在一个站点比较了宏条形码和形态学方法,以优化甲藻的鉴定和检测。结果显示,在分类群的深度特异性相对丰度上,甲藻群落的分层明显;5~30m与130~150m差异最大。甲藻纲(光合和异养)的相对丰度随深度的增加而降低,而共甲藻目(寄生)的相对丰度随深度的增加而增加。各站主要类群组成相似。ASVs的物种丰富度和多样性在不同深度和站点之间相似;但优势类群丰度在0~30m最高,稀有类群丰度在130~150m最高,对特定深度的地层具有一定的适应能力。在科和种水平上,未分类的ASVs数量较多,尤其是以Syndinian为代表的。总之,珊瑚海开放水域的鞭毛藻群落具有高度的多样性,并随深度而分层;沿深度梯度的相对丰度分布模式反映了环境因子和生态过程。与传统的显微镜分析方法相比,宏条形码检测出更多的物种丰富度,但两种方法是互补的,形态学分析显示出更多的物种丰富度。

图3 不同站点不同深度的甲藻ASVs的相对丰度
以上就是今天给大家分享的关于eDNA宏条形码在水体中的应用。通过以上案例,也不难发现同为水体样本,不同的引物可以检测不同的目标物种。若想获得更丰富的物种,建议选择多对引物进行检测。
参考文献
[1] Chen X, Li S, Zhao J, et al. Passive eDNA sampling facilitates biodiversity monitoring and rare species detection[J]. Environment International, 2024, 187: 108706.
[2] Cerrillo-Espinosa P, Calderón-Aguilera L E, Medina-Rosas P, et al. eDNA metabarcoding shows highly diverse but distinct shallow, mid-water, and deep-water eukaryotic communities within a marine biodiversity hotspot[J]. bioRxiv, 2024: 2024.07. 14.603441.
[3] Carve M, Manning T, Mouradov A, et al. eDNA metabarcoding reveals biodiversity and depth stratification patterns of dinoflagellate assemblages within the epipelagic zone of the western Coral Sea[J]. BMC Ecology and Evolution, 2024, 24(1): 38.


